
The NGS® hydroponic system features the following advantages: · Flexible, customizable system · Water savings · Labor savings · High density per Ha · Greater. I-NGS Aurora App wuhlelo lokusebenza lwakamuva lwe-NGS lwe-smartphone, eklanyelwe ukuphatha uhambo oludidiyelwe kanye nesixazululo sokuphendula izimo. Puedes configurarlas en base a tus preferencias en nuestro apartado CONFIGURAR OPCIONES: toma el control y disfruta de una navegación personalizada en nuestra.
Contenido del artículo:
S.a.i Ngs 900va 360w 2xschuko Negra (FORTRESS900V3)
Boulevard du 11 novembre num43 Villeurbanne cedex Ver en el mapa. Given I n g s inherent dynamics of a viral quasispecies, w are often interested I n g s the comparison of diversity indices of sequential samples of a patient, or in the comparison of diversity indices of virus in groups of patients in a treated versus control design. Incubar durante 10 min a TA. Hipomagnesemia primaria autosómica dominante con hipocalciuria.



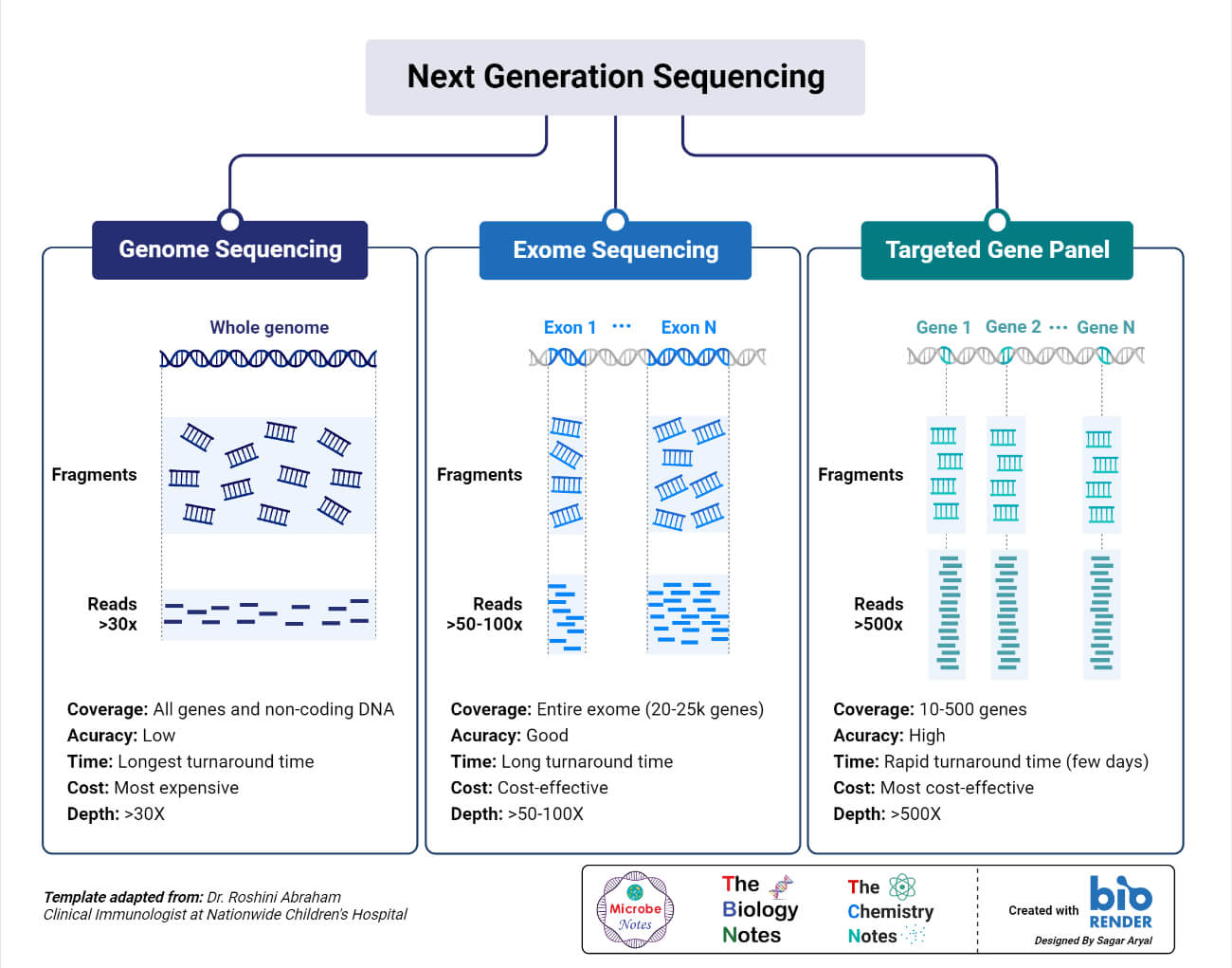
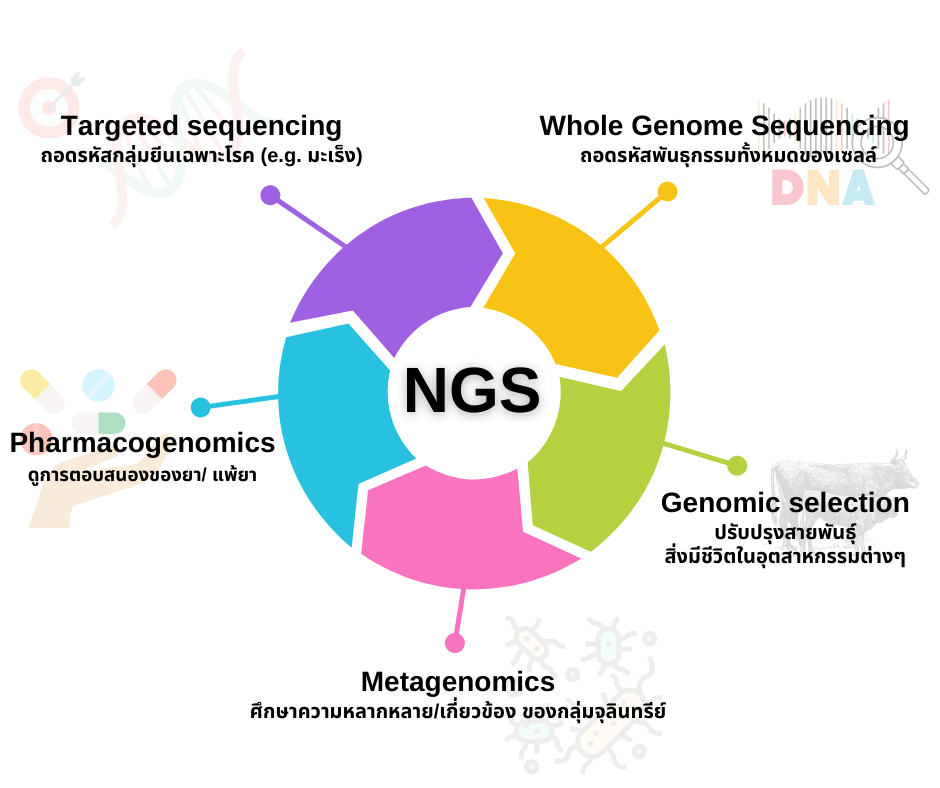
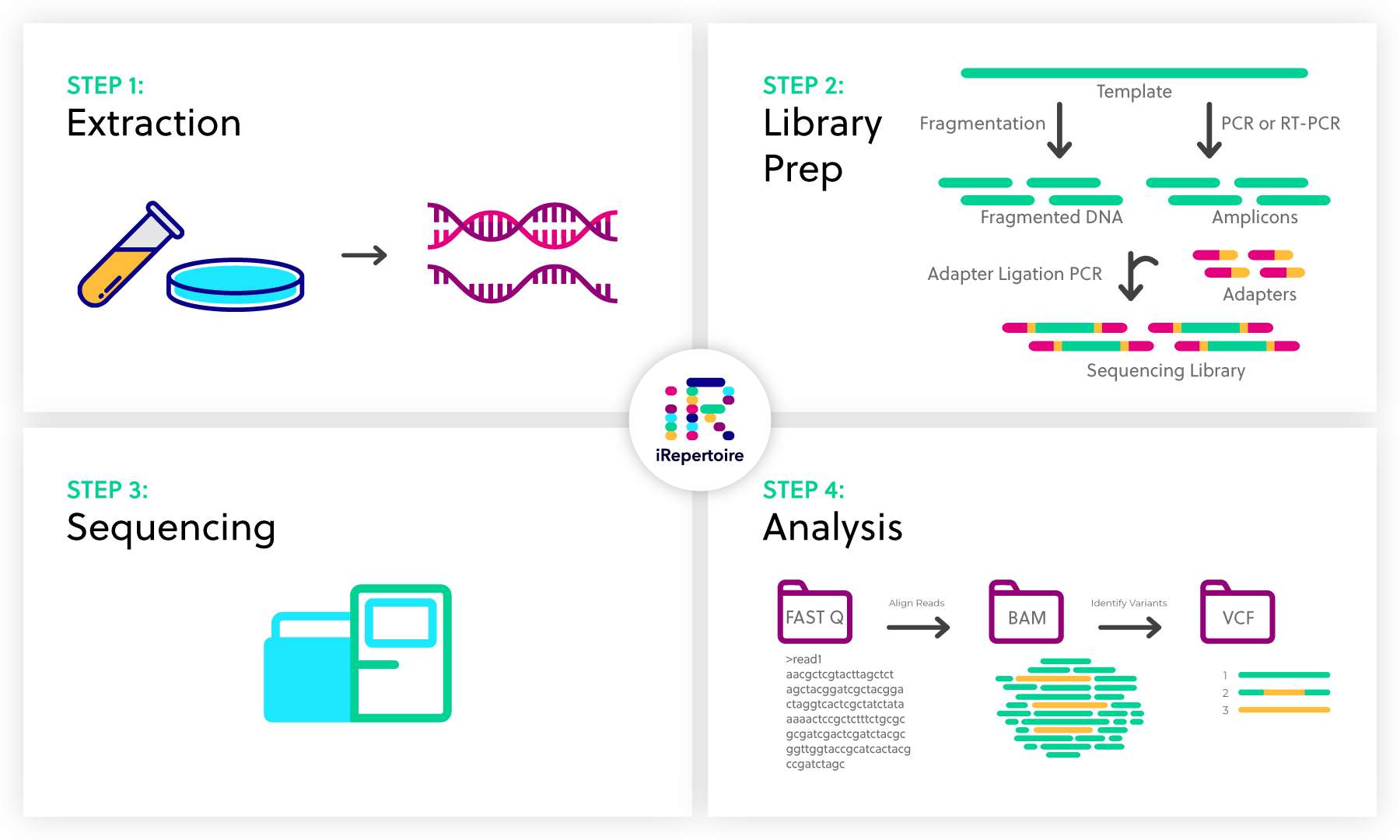
La secuenciación paralela masiva es cualquiera de varios enfoques de alto rendimiento para la secuenciación de ADN que utilizan el concepto de procesamiento paralelo masivo; también se denomina secuenciación de próxima generación NGS, por sus siglas en inglés Next-Generation Sequencing o secuenciación de segunda generación.
Algunas de estas tecnologías aparecieron entre y y han sido comercialmente accesibles desde Muchas plataformas NGS difieren en los diseños de ingeniería y la química de secuenciación. Ambos utilizan plantillas de ADN amplificadas por clonación y espacialmente separadas o moléculas de ADN individuales en una celda de flujo para realizar una secuenciación paralela masiva.
Los métodos basados en síntesis cometen menos errores de secuenciación, pero no permiten obtener información extra de la secuencia secuenciada modificación de bases como por ejemplo la metilación. Los métodos no basados en síntesis ejemplo, Oxford NanoPore , permiten analizar secuencias de ADN, o incluso ARN, y analizar las modificaciones de las bases de esa secuencia.
Los métodos de lectura corta tienen una capacidad enorme de producción de datos, pudiendo secuenciar genomas enteros en muy poco tiempo en algunos casos, pero no pueden saber si varios fragmentos de ADN secuenciados provienen de la misma fuente.
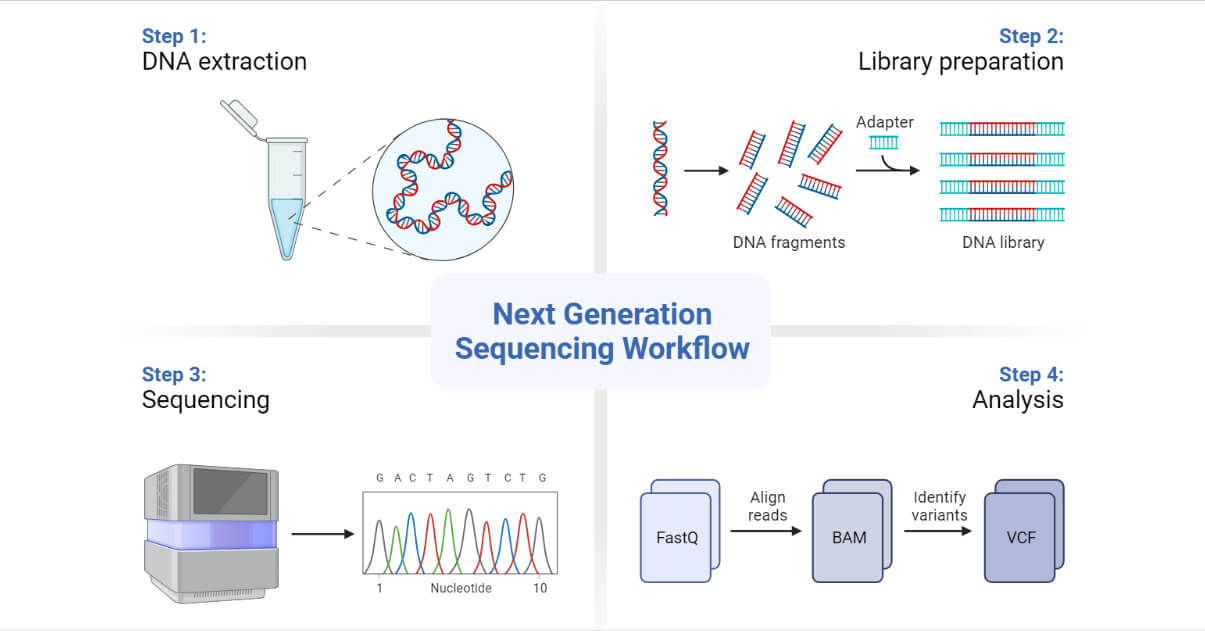
Un ejemplo de este tipo de tecnología sería Illumina. PacBio y Oxford NanoPore serían ejemplos de tecnologías de lectura larga una de síntesis y la otra de no síntesis, respectivamente. En general, la secuenciación del ADN mediante síntesis se lleva a cabo utilizando plataformas NGS disponibles comercialmente siguiendo los pasos que se enumeran a continuación. En primer lugar, se utiliza la amplificación clonal in vitro mediante PCR para crear bibliotecas de secuenciación de ADN.
En segundo lugar, en lugar de usar la química de terminación de cadena para identificar la secuencia de ADN, el ADN se secuencia mediante síntesis, en cuyo caso se agregan nucleótidos a la cadena complementaria. La mayoría de las plataformas NGS de síntesis siguen estos pasos, pero cada una lo hace de manera diferente.
En una ejecución de un solo instrumento, la paralelización NGS de las reacciones de secuenciación produce cientos de Megabases Mb a Gigabases Gb de lecturas de secuencias de nucleótidos. En la tabla se resumen las plataformas de secuenciación masiva en paralelo disponibles para el comercio a partir de y sus características.
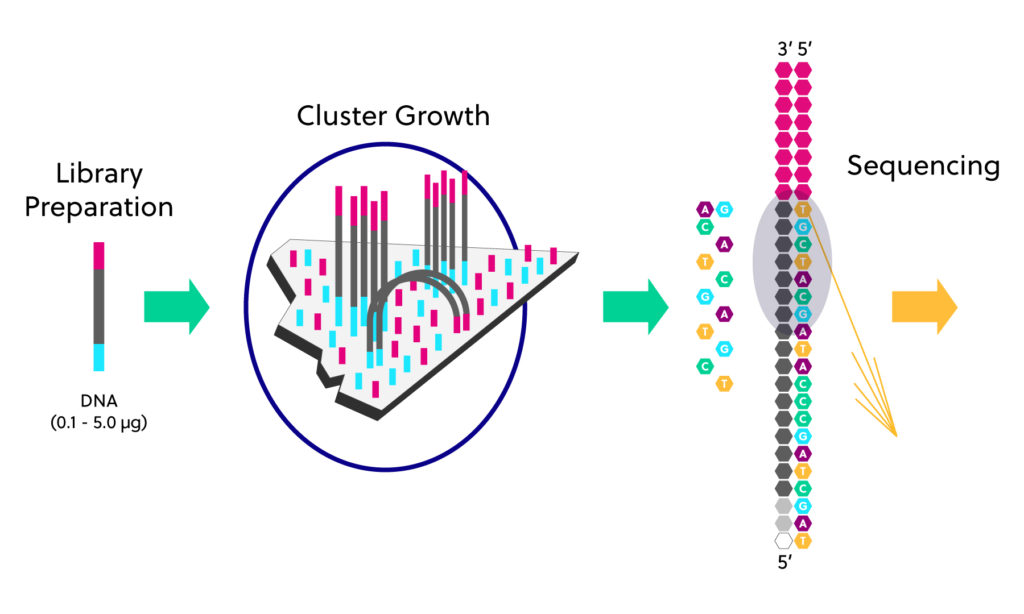
Se indican los tiempos de ejecución y la salida de Gigabase Gb por ejecución para la secuenciación de un solo extremo. Los tiempos de ejecución y las salidas se duplican aproximadamente cuando se realiza una secuenciación de extremos emparejados. Se utilizan dos métodos para preparar plantillas para reacciones de NGS: plantillas de moléculas de ADN individuales y plantillas amplificadas que se originan a partir de moléculas de ADN individuales.
La distribución final de las plantillas puede ser espacialmente aleatoria o en cuadrícula. Los fragmentos de ADN monocatenario plantillas se unen a la superficie de las perlas con adaptadores o enlazadores, y una perla se une a un solo fragmento de ADN de la biblioteca de ADN. La superficie de las perlas contiene sondas de oligonucleótidos con secuencias que son complementarias a los adaptadores que unen los fragmentos de ADN.
Next Generation Sequencing 1: Overview - Eric Chow (UCSF)




Deja un comentario